[ para seguir la introducción:
1, 2]
Introducción
Por 'familias (o subfamilias) de proteínas' nos referimos
a grupos de secuencias procedentes de varias especies y que realizan una
función similar y tienen un mismo origen evolutivo. Sin embargo,
no todas las proteínas relacionadas evolutivamente pertenecen a
una misma familia y a veces realizan funciones muy distintas. El parecido
entre las proteínas de una familia y otra es menor que entre las
proteínas de una misma familia, siendo estos valores de parecido
muy variables según la familia de que se trate (puede darse el caso,
como veremos, de que el parecido entre dos familias no sea fácilmente
reconocible). Teniendo en cuenta esto podemos pensar que el 'espacio de
secuencias' está formado por 'islas' (familias) y la distancia entre
éstas depende de si tienen una relación evolutiva o no y,
si la tienen, de cuán parecidas son.
Otro concepto importante que nos será útil para realizar
esta práctica es el de que la homología es transitiva.
Decimos que hay una relación de homología si hay un parecido
que demuestra un origen evolutivo común. Que la homología
es transitiva quiere decir que si una proteína A es homóloga
a otra B, y B es homóloga a C, entonces A es homóloga a C,
aunque no se parezcan. Sin embargo, las proteínas están constituidas
por dominios que aparecen en múltiples combinaciones y esto puede
hacer que la transitividad no sea aplicable; podemos matizar así:
la homología es transitiva a nivel de dominios.
PROTOMAP
es una base de datos en la que (casi) todas las proteínas conocidas
están agrupadas en 'clusters' mediante un algoritmo que trata
de detectar automáticamente los límites entre familias y
las distancias entre ellas. Hablaremos de 'clusters' y no 'familias'
porque a menudo un cluster se corresponde con una subfamilia, o contiene
varias familias (no es perfecto!).
Existen distintos niveles de exigencia para decidir si un cluster ha
de contener proteínas más o menos próximas. Nosotros
trabajaremos al nivel menos restrictivo, el de 1e-00 (1) pues es el más
adecuado para estudiar grandes distancias evolutivas.
Siempre que entremos en un cluster podremos ver (entre otras cosas):
1) qué proteínas forman parte de él: su función,
su origen (arqueas, bacterias, eucariotas, virus...)
2) un 'link' para ver qué clusters hay cercanos al cluster en que nos encontramos.
Cuando pinchemos en ese link ('possibly related clusters') veremos una lista de clusters, donde se indicará cuántas proteínas contiene ese cluster, qué 'distancia' y cuántas conexiones hay respecto al cluster en que estamos. Protomap toma como relación cualquier parecido de secuencia (mediante BLAST, por ejemplo) con un E-value menor de 1. Estos e-values son poco fiables, pero si vemos que hay muchos de estos e-values, la confianza será mayor.¡Empecemos!
Estas son sus secuencias:
>RASH_HUMAN|P01112|Transforming protein P21/H-RAS-1 (C-H-RAS).
MTEYKLVVVG AGGVGKSALT IQLIQNHFVD EYDPTIEDSY RKQVVIDGET CLLDILDTAG
QEEYSAMRDQ YMRTGEGFLC VFAINNTKSF EDIHQYREQI KRVKDSDDVP MVLVGNKCDL
AARTVESRQA QDLARSYGIP YIETSAKTRQ GVEDAFYTLV REIRQHKLRK LNPPDESGPG
CMSCKCVLS
>EFTU_ECOLI|P02990|Elongation factor Tu (EF-Tu) (P-43).
SKEKFERTKP HVNVGTIGHV DHGKTTLTAA ITTVLAKTYG GAARAFDQID NAPEEKARGI
TINTSHVEYD TPTRHYAHVD CPGHADYVKN MITGAAQMDG AILVVAATDG PMPQTREHIL
LGRQVGVPYI IVFLNKCDMV DDEELLELVE MEVRELLSQY DFPGDDTPIV RGSALKALEG
DAEWEAKILE LAGFLDSYIP EPERAIDKPF LLPIEDVFSI SGRGTVVTGR VERGIIKVGE
EVEIVGIKET QKSTCTGVEM FRKLLDEGRA GENVGVLLRG IKREEIERGQ VLAKPGTIKP
HTKFESEVYI LSKDEGGRHT PFFKGYRPQF YFRTTDVTGT IELPEGVEMV MPGDNIKMVV
TLIHPIAMDD GLRFAIREGG RTVGAGVVAK VLS
Si queréis podéis probar con BLAST para determinar esta cuestión. Pero os adelanto que el mejor alineamiento local que obtendréis será muy pequeño y poco significativo (=no distinguible de un parecido al azar):
ras : ESRQAQDLARSYGIPYI
eftu: QTREHILLGRQVGVPYI
Sugerencia de pasos a seguir:
1.- Encuentra a qué cluster pertenece la proteína ras/p21 de humanos (accession: rash_human). Haz click en 'access/search the hierarchy of clusters'. En la parte de arriba de la página que aparecerá, selecciona la búsqueda por 'Swissprot ID/AC', introduce el nombre 'rash_human' y dale a 'search'. Te aparecerá esto: la lista de clusters con las que tiene alguna 'conexión' (BLAST-hit con E-value < 1). El primer cluster que aparece es el suyo propio ('main cluster') y es el 18. Pinchad en él y veréis esto. ¿Qué proteínas están presentes en ese cluster? Si vas bien de tiempo, echa un vistazo a sus funciones y di si piensas que en el cluster hay una subfamilia o más.
2.- Pincha en 'possibly related clusters' para ver quiénes son sus vecinos (veréis esto). Id pinchando en los distintos clusters (fijaos sólo en los grandes, así ahorraréis tiempo). ¿Encontráis algún cluster que contenga el EF-Tu de E.coli? Sí?, perfecto. No? probad a identificar a qué cluster pertenece el EF-Tu de E.coli tal y como hicisteis para identificar el de rash_human y una vez sepáis qué número de cluster tiene, buscadlo en la lista de clusters vecinos del cluster de ras. El cluster de EF-Tu es el 19!b) ¿Con qué otros factores implicados en la traducción de proteínas podéis decir que ras está relacionada? ¿Con los IF-2?
¿Cuántas conexiones hay entre ambos clusters y cuán significativas son (e-value)?
¿Creéis que ambas familias están relacionadas evolutivamente?
Pinchad en el cluster de EF-Tu (el 19), ¿qué otras proteínas están en ese grupo?
¿Ya lo has encontrado? ¿Qué clusters dirías que están más cercanos: IF-2 (cluster 434), EF-Tuo (cluster 19) RAS (cluster 18)
c) Finalmente, y más difícil, ¿está
relacionada evolutivamente la proteína rash_human con PPS2_HUMAN?
PPS2_HUMAN es una 'Bifunctional 3'-phosphoadenosine 5'-phosphosulfate synthethase' y no une GTP como las otras que hemos visto, sino ATP.
Pista: este cluster no esta relacionado directamente con el de ras, aplicad la transitividad entre clusters (teniendo en cuenta que la transitividad sólo vale cuando las regiones similares se corresponden). Para ver qué regiones son las implicadas en los parecidos de secuencia pinchad en 'List of connections (and alignments)' (veréis esto) y luego pinchad el botón de 'See alignments'.
Otra pista: el cluster de PPS2_HUMAN es el 740.
¿Qué conclusión podéis sacar?
Por último, decir que estas cuestiones también se podrían haber resuelto de otras formas, como por ejemplo, utilizando perfiles o HMMs, los cuales permiten determinar relaciones evolutivas distantes porque incorporan información precisa de la familia en cuestión, como por ejemplo que los residuos X e Y sean un Trp y una Lys, dando más importancia a la conservación de éstos que a la de otros residuos.
Aquí tenéis la prueba de que el parecido se debe
a un mismo origen evolutivo: las proteínas tienen una misma estructura
3D, y pertenecen a la superfamilia ' P-loop
containing nucleotide triphosphate hydrolases', según SCOP.
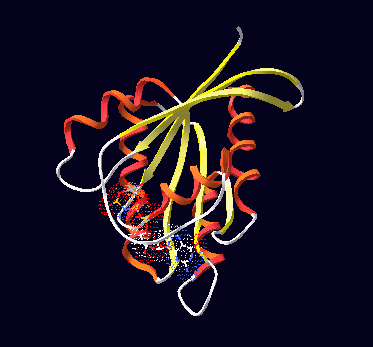
3D structure of human ras/p21 (swissprot: rash_human; pdb: 1ctq) |
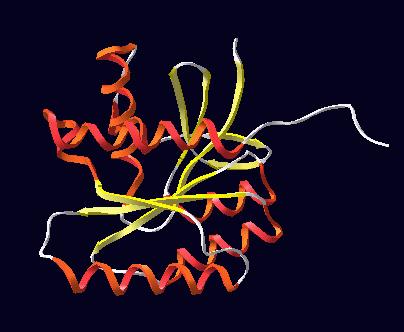
3D structure of E. coli EF-TU GTP-binding domain (swissprot: eftu_ecoli; pdb: 1dg1) |
|
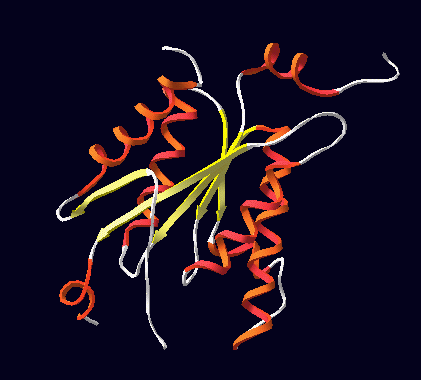
3D structure of Adenylylsulfate kinase ATP-binding domain (swissprot: kaps_pench; pdb: 1d6j) |
||
![]()
| Protein Design
Group.
Centro Nacional de Biotecnología (CNB - CSIC). Campus Universidad Autónoma de Madrid. Cantoblanco. 28049 Madrid. Spain. Phone:+34-91-585 46 69 Fax: +34-91-585 45 06 |
Federico
Abascal
e-mail: fabascal@cnb.uam.es Last update: April, 2002 |