 |
Bioinformatics Unit - CNIO |
|
Práctica de clustering para datos de DNA arrays |
|
 |
Bioinformatics Unit - CNIO |
|
Práctica de clustering para datos de DNA arrays |
|



Estos datos son los correspondientes a un experimento hecho por un grupo de Stanford sobre el salto diauxico (paso de condiciones aerobias a condiciones anaerobias en S. cerevisiae)
Los datos consisten en una tabla donde aparecen los valores en columnas separadas por tabuladores. La 1a columna corresponde al nombre de la ORF, la 2a al del gen si este se conoce y a partir de la 3a encontramos los datos experimentales.
En este caso, también nos dan los resultados de la división, así que nos basta con extraerlos de la tabla.
Ahora aplicamos una transformación logarítmica a los datos (base 2).
He aquí el árbol correspondiente a estos datos, usando el UPGMA y midiendo correlación lineal:
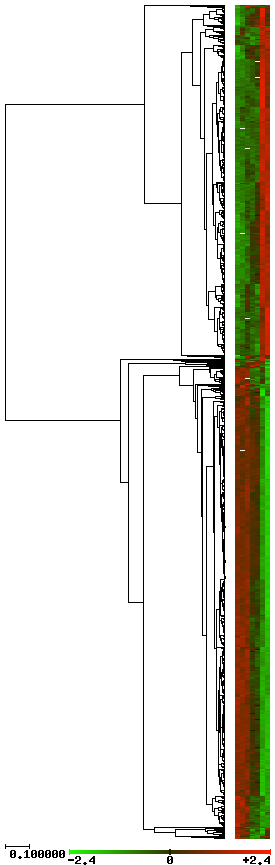
Aparecen dibujados en el margen los patrones correspondientes a cada gen en una escala de colores (ver pie de la imagen).
Y lo mismo, pero usando el SOTA y con un nivel de confianza en la variabilidad del 90%.
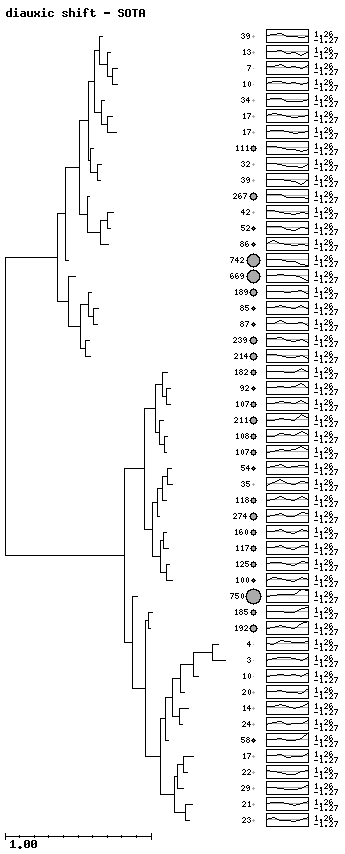
En este caso, se representa con un círculo más o menos grande, el tamaño de los clusters (el número de genes que tiene). Al lado aparece el patrón del nodo correspondiente.
La variabilidad se define en cada cluster como la mayor de las distancias entre dos patrones asignados a ese cluster, esto es, los dos patrones más alejados en un grupo definen su variabilidad. El nivel de confianza se basa en un método estadístico: se barajan todos los datos (en columnas y filas) y se calcula la distribución de distancias (distancias aleatorias); se corta esta distribución al nivel de confianza deseado y se toma la distancia correspondiente como umbral para parar el clustering.
Por último, podemos ver el resultado de aplicar el SOM a estos mismos datos. Lo cierto es que el SOM sólo acepta distancias euclídeas por lo que es necesario estandarizar los patrones previamente, pero antes de poder estandarizar los patrones es imprescindible eliminar aquellos para los que la relación señal / ruido sea demasiado baja.
Eliminamos los datos que no sean relevantes, esto es, los que no varían significativamente. En concreto, se han eliminado los que no varían más de dos veces en al menos dos de los puntos medidos. Puesto que tenemos los datos en una escala logarítmica, tienen que tener al menos dos valores por encima de 1 o por debajo de -1.
Finalmente, estandarizamos los patrones:
Ahora ya podemos aplicar el SOM y este es el resultado:
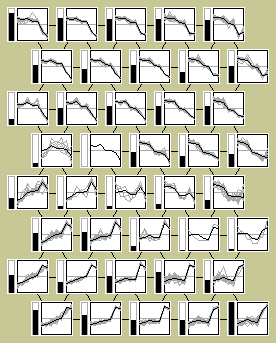
Cada recuadro corresponde a una de las neuronas del mapa. En cada neurona se representa con trazo negro grueso el perfil de la neurona y el trazo gris fino los perfiles de cada uno de los patrones de expresión asignados a esa neurona. La altura de las barras negras es directamente proporcional al número de patrones asignados al nodo.
| CNIO, Unidad de Bioinformática. Abril de 2002 |