| volver al índice |
Análisis de secuencias:
motivos y perfiles
Parte teórica

Por ejemplo, si dos proteínas se parecen en un 15 o un 20% de su secuencia (es decir, sólo coinciden en uno de cada 5 ó 6 aminoácidos) no sabremos si son homólogas, pero si resulta que este parecido se concentra en los residuos funcionales de la proteína (p.e. en el centro activo), la probabilidad de que tengan un origen evolutivo común es mayor. ¿Cómo podemos saber si las identidades se concentran en esos residuos importantes? Es lo que veremos hoy. La fuente principal son los alineamientos múltiples.
Estos aspectos son los que han motivado el surgimiento de métodos de comparación de secuencias (y de búsqueda de homólogos) más sofisticados, entre los que destacamos los patrones, los perfiles y los HMMs (hidden markov models o modelos de Markov ocultos).
En
esta clase explicaremos estos métodos y también los recursos
que podemos encontrar en la web para utilizarlos. Asimismo, veremos que
existen muchas bases de datos que utilizan estos métodos y ofrecen
mucha información. Posteriormente hablaremos de las familias de
proteínas, del interés que tiene su clasificación
y estudio y de cómo los perfiles y otros métodos nos ayudan
a identificar las familias dentro del océano de secuencias
que conocemos.
Secuencias consenso y expresiones regulares
Cuando consultamos la literatura referente a estos aspectos que vamos a tratar de las proteínas, encontramos que existe cierta laxitud y, a veces también mal uso, respecto a algunos términos como son "motivo" o "dominio". El segundo, el término dominio, lo introduciremos más adelante.
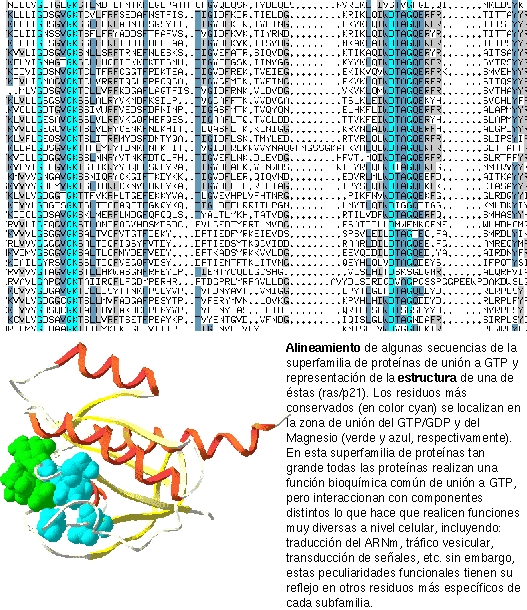
Motivo: si observamos un alineamiento múltiple de proteínas homólogas veremos que algunas columnas varían bastante, mientras que otras están más conservadas. Cuando observamos ciertas columnas cercanas con una alta conservación, es decir, cuando encontramos trocitos de las secuencias que se conservan más que otros y que podrían caracterizar funcionalmente a las proteínas, entonces solemos hablar de MOTIVOS. (ver ejemplo en el alineamiento de más arriba)
Los alineamientos múltiples son la fuente principal para determinar qué partes de la secuencia son más importantes para su función o estructura, y existen diversas aproximaciones para utilizar esta información.
AGTVATVSCAGTSATHACIGRCARGSCIGEMARLACIGDYARWSC.........IGTVARVSC <= Ejemplo de secuencia consenso
Estas expresiones o patrones se pueden utilizar para caracterizar motivos, indicando qué posiciones son más importantes y cuáles pueden variar y qué variaciones pueden sufrir. Primero veremos cómo hablar en el lenguaje de las expresiones regulares.
[AC]-x-V-x(4)-{ED}
Este patrón significa: [Ala o Cys]-cualquiera-Val-cualquiera-cualquiera-cualquiera-cualquiera-{cualquier aa excepto Glu y Asp}< A-x-[ST](2)-x(0,1)-V
Este patrón debe encontrarse en posición N-terminal ('<') y significa: Ala-cualquiera-[Ser o Thr]-[Ser o Thr]-(un o ningún aminoácido de cualquier tipo)-Val<{C}*>
Este patrón lo cumplen todas aquellas proteínas que no contienen cisteínas. El * significa 'cero o más elementos'.
AGTVATVSC
AGTSATHAC
IGRCARGSC
IGEMARLAC
IGDYARWSC
.........
IGTVARVSC <= Ejemplo de secuencia consenso
[AI]-G-X-X-A-[RT]-[SA]-C
Primeramente definiremos el concepto de dominio, ya que tiene bastante relación con los perfiles, los cuales usualmente cubren una mayor parte de las secuencias que los motivos. Este concepto de dominio se utiliza con cierta flexibilidad pero generalmente define una unidad estructural independiente. Sin embargo, en estudios genéticos de deleción a veces se utiliza como sinónimo de la parte mínima de la secuencia capaz de realizar la función estudiada. En las bases de datos de dominios como PFam, un dominio se suele corresponder con el núcleo del dominio estructural, aquella zona más similar entre todas las proteínas de una familia, aunque no tiene por qué coincidir exactamente con los límites del dominio estructural.
Un perfil es una matriz de sustitución específica para cada posición de la secuencia (position specific scoring matrix). Esta matriz, en lugar de tener como dimensiones 20x20, tiene como dimensiones 20xL, siendo L la longitud del alineamiento múltiple. A partir del alineamiento múltiple se construye dicha matriz teniendo en cuenta la frecuencia de los aminoácidos en cada posición así como sus propiedades físico-químicas (es decir, combina la información del alineamiento múltiple con la información de una matriz del tipo de BLOSUM).
Existen distintos métodos para construir estas matrices. El método clásico es el de Gribskov y colaboradores. Es el método que por ejemplo se emplea en PROSITE (donde además de patrones se utilizan perfiles para describir algunas familias), aunque con algunas modificaciones.
A
continuación se muestra un alineamiento múltiple y cómo
se construiría el perfil según el método empleado
en PROSITE.
F K L L S H
C L L V
F K A F G Q
T M F Q
Y P I V G Q
E L L G
F P V V K E
A I L K
F K V L A A
V I A D
L E F I S E
C I I Q
F K L L G N
V L V C
A -18 -10 -1 -8 8 -3 3 -10 -2 -8
Como se puede apreciar, los pesos para cada aminoácido para cada columna no sólo se corresponden con sus frecuencias, sino también con sus propiedades físico-químicas, ya que se utiliza la información contenida en matrices como BLOSUM62 para completar la información. Por ejemplo, la A (alanina) de la tercera columna recibe una puntuación menor (-1) que la M (metionina) (+10), a pesar de que no haya ninguna metionina. Esto es porque la M es más parecida en sus propiedades a L, I, V y F que la alanina.
Los perfiles también son capaces de incluir información de inserciones y deleciones.
Alineamiento de una secuencia con un perfil:
S E Q U E N
C E
. . . . . .
. . .
p
. . . . . .
. . .
r \
. . _ . _ . . . .
. .
o
\
. . . . . .
. . .
f
\
. . . . . .
. . .
i
|
. . . . . .
. . .
l
\
. . . . . .
. . .
e
. . . . . .
. . .
Es similar al alineamiento de dos secuencias, pero no se comparan pares de aminoácidos o posiciones sino que cada aminoácido de la secuencia se compara con cada posición del perfil. Es decir, la puntuación no se obtiene de una matriz como BLOSUM, sino que viene implícita en el perfil con el que estamos alineando nuestra secuencia.
El
camino definido mediante las líneas horizontales, verticales y diagonales
describe el alineamiento:
S E Q U E - Nr - - o f i l
A
-18 -10 -1 -8 8 -3 3 -10
-2 -8
C
-22 -33 -18 -18 -22 -26 22 -24 -19 -7
D
-35 0 -32 -33 -7 6 -17 -34 -31
0
E
-27 15 -25 -26 -9 23 -9 -24 -23 -1
F
60 -30 12 14 -26 -29 -15 4 12 -29
G
-30 -20 -28 -32 28 -14 -23 -33 -27 -5
H
-13 -12 -25 -25 -16 14 -22 -22 -23 -10
I
3 -27 21 25 -29 -23 -8 33 19 -23
K
-26 25 -25 -27 -6 4 -15 -27 -26 0
L
14 -28 19 27 -27 -20 -9 33 26 -21
M
3 -15 10 14 -17 -10 -9 25 12 -11
N
-22 -6 -24 -27 1 8 -15 -24 -24 -4
P
-30 24 -26 -28 -14 -10 -22 -24 -26 -18
Q
-32 5 -25 -26 -9 24 -16 -17 -23 7
R
-18 9 -22 -22 -10 0 -18 -23 -22 -4
S
-22 -8 -16 -21 11 2 -1 -24 -19 -4
T
-10 -10 -6 -7 -5 -8 2 -10 -7
-11
V
0 -25 22 25 -19 -26 6 19 16 -16
W
9 -25 -18 -19 -25 -27 -34 -20 -17 -28
Y
34 -18 -1 1 -23 -12 -19 0 0 -18
supongamos que queremos alinear la secuencia FKTLGCCLLV:
El mejor alineamiento será:
F K L L S H
C L L V
F K A F G Q
T M F Q
Y P I V G Q
E L L G
F P V V K E
A I L K
F K V L A A
V I A D
L E F I S E
C I I Q
F K L L G N
V L V C
F K T L G
C C L L V
Y la puntuación: 60 25 -6 27
28 -26 22 33 26 -16
lo cual suma en total: 173.
Éste es un caso sencillo, sin incluir 'gaps', es decir, sin inserciones ni deleciones.
Cada puntuación tiene un significado estadístico, es decir, se pueden obtener p-values o e-values.
Los perfiles sin embargo son sensibles a determinados problemas. Por ejemplo, si existe un sesgo en la representación de las secuencias del alineamiento, es decir, si hay muchas secuencias demasiado parecidas y sólo unas pocas un poco más divergentes, entonces el perfil dará preferencia a encontrar secuencias del primer tipo. Existen formas de corregir estos sesgos, por ejemplo dando distintos pesos a las secuencias.
Por otra parte, en algunos casos es mejor usar patrones, especialmente cuando queremos describir motivos pequeños en los que no debería haber variaciones, por ejemplo, sabemos que en dicho motivo en la posición x debe haber un glutamato (E) y como tal lo expresamos en la expresión regular. Sin embargo, la flexibilidad del perfil podría tolerar que en esa posición un aspártico (D) obtuviera una puntuación positiva (E y D son muy parecidos en sus propiedades físico-químicas).
La limitación más importante
de los perfiles es que el sistema de puntuación que emplean tiene
una base estadística pobre.
Los modelos de Markov ocultos (HMMs: hidden Markov models) se desarrollaron inicialmente para el reconocimiento automático de voz. Este problema es similar al del reconocimiento de relaciones evolutivas entre las proteínas. Lo que se hacía era fragmentar el mensaje hablado en trocitos (frames) o sonidos aislados (de 10-20 milisegundos). Cada trocito o frame era automáticamente asignado a una de 256 categorías de sonido predefinidas (a la que más se pareciera). El resultado de esto era una cadena o larga secuencia de etiquetas de categorías a partir de las cuales realizar el reconocimiento de voz automático, detectando qué fonemas y qué palabras se han dicho. El problema es que existen grandes variaciones en la pronunciación, también en la duración de las distintas partes de la palabra.
Un HMM se entrena a partir de diversas observaciones en las que esperamos que las posibles variaciones se hayan producido, por ejemplo, para reconocer la palabra "vaca" u "otorrinolaringólogo" se debe entrentar el HMM a partir de muchas muestras de "vaca" y "otorrinolaringólogo", pronunciadas en distintos contextos o por distintas personas. El resultado es una estructura del siguiente modo:
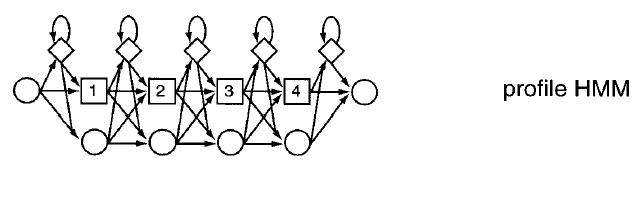
Un HMM está compuesto por una serie de nodos o estados cada uno de los cuales emite símbolos (una de las 256 categorías de sonido o uno de los 20 aminoácidos, por ejemplo) con una probabilidad dada (¡como en los perfiles!). Los distintos estados están conectados ¡secuencialmente! existiendo probabilidades de transición entre ellos. Además existen probabilidades de inserción y deleción (en realidad éstas también se consideran estados). Eso es lo que son básicamente los HMMs, cuya principal ventaja es que tienen una base probabilística muy sólida.
Una vez entrenado un HMM para "vaca", cada vez que alguien diga algo y lo convirtamos en una cadena de etiquetas podremos determinar con qué probabilidad esa cadena podría ser emitida por el HMM de "vaca". En el caso de las secuencias lo tenemos más fácil incluso, ya que éstas ya vienen en forma de cadenas o secuencias.
Un ejemplo:
Por último señalar que éste es sólo un ejemplo del amplio abanico de aplicación de los HMMs, que pueden construirse con muy variadas arquitecturas y aplicarse para solucionar muy diversos problemas:
Búsquedas con secuencias intermedias (ISS: intermediate sequence searches)
Este método no utiliza información de qué posiciones son más importantes, ni utiliza alineamientos múltiples. Sin embargo, puede superar algunas de las limitaciones que tienen los métodos de búsquedas simples (BLAST, FASTA, ...).
Recordemos la limitación de BLAST: que no es capaz de distinguir entre parecidos que son reflejo de una relación de homología y parecidos que se producen al azar cuando el % de identidad está por debajo del ~25% (aprox.).
¿Cómo consiguen las búsquedas con secuencias intermedias
superar esta limitación? Gracias a que la homología entre
las proteínas presenta la propiedad transitiva, esto es:
si la proteína A es homóloga a la proteína B, y B
es homóloga a C, entonces A también es homóloga a
C (aunque A y C no se parezcan). Esto sólo es cierto cuando las
zonas homólogas se corresponden, es decir, sólo se aplica
a nivel de dominios.
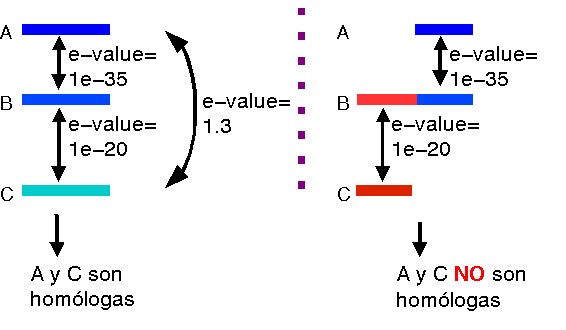
Por tanto, si realizamos búsquedas recursivas con los homólogos que vamos encontrando quizás seamos capaces de encontrar homólogos remotos, pero su éxito depende de que existan secuencias intermedias a distancias suficientemente cercanas (por ejemplo con % de identidad de secuencia > 30%). El método es como ir saltando de secuencia en secuencia. Más adelante veremos que, además de la detección de homólogos remotos, este método tiene otras utilidades para clasificar las proteínas en familias.
Ejemplo:

Algunas bases de datos de patrones, perfiles y hmms. Prosite y Pfam.
A continuación
describiremos las bases de datos PROSITE y PFam, de forma que podamos realizar
la práctica con ellas. En la siguiente lección hablaremos
de las familias de proteínas y en ese contexto volveremos a hablar
de esas bases de datos y de otras similares.
La versión 17.39, del 06 de marzo de 2003, contiene 1173 entradas de documentación, las cuales describen 1609 patrones, reglas (rules) y perfiles. En http://us.expasy.org/prosite/prosuser.html hay una descripción de la base de datos.
Y en http://us.expasy.org/cgi-bin/nicedoc.pl?PDOC50020 hay un ejemplo de una entrada de documentación.
La construcción de los patrones es manual: a partir de revisiones bibliográficas acerca de familias de proteínas, se consultan los alineamientos múltiples y se derivan expresiones regulares. La eficiencia (sensibilidad/especificidad) de estos patrones es comprobada aplicándolos a las secuencias de la base de datos Swiss-Prot (la cual está anotada por expertos). Si el patrón no es satisfactorio se intenta refinar. En este procedimiento se intenta generar patrones los más cortos posibles.
En Prosite también existen perfiles. Se construyen a partir de alineamientos múltiples.
Existen dos tipos de entradas en Prosite: las que definen los patrones y los perfiles y las que contienen la documentación.
La estructura de una entrada de Prosite es ésta:
ID Identification (Begins each entry; 1 per entry)
'A': archaea'B': bacteriofagos'E': eucariotas'P': procariotas o bacterias'V': virus de eucariotas
(hay más información del formato de PROSITE aquí).
Tour: Prosite:
(browse,
search,
scanprosite,
motifscan,
pratt)
Pfam (http://www.sanger.ac.uk/Software/Pfam/index.shtml) es una base de datos de perfiles tipo HMM. Se divide en dos partes: pfam-A y pfam-B. La primera se construye manualmente: cada vez que se identifica una nueva familia de proteínas un experto elabora un HMM diagnóstico: un HMM capaz de detectar a todas las otras proteínas de la familia y sólo a éstas. Por otra parte, como pfam-A sólo cubre el 73% de Swiss-Prot y TrEMBL, existe pfam-B. Ésta se genera automáticamente a partir de aquellos perfiles que existen en PRODOM (también generados automáticamente) que no se corresponden con ningún pfam-A. Un 20% de las proteínas de Swiss-Prot y TrEMBL presentan al menos un pfam-B.

Pfam-A contiene 5193 familias de proteínas (bastante más que Prosite).
Pfam trabaja con dominios, es decir, cada perfil HMM se corresponde con un dominio, aunque no necesariamente cumplan la definición de dominio estructural independiente, sino más bien suelen ser regiones características de una determinada familia de proteínas.
Además de las ventajas que de por sí tiene esta clasificación, pfam resulta útil para:
-analizar los alineamientos múltiples que contiene.Tour: Pfam: (browse, protein search, dna search, taxonomy). En el ejemplo de CARD (http://www.sanger.ac.uk/cgi-bin/Pfam/getacc?PF00619) podemos ver algunas de las características de una entrada de Pfam.
-estudiar la organización de dominios de las proteínas.-examinar la distribución filogenética de las proteínas que presentan el dominio.
-también permite ver la estructura tridimensional de los dominios, cuando ésta se conoce.
-y, de las más importantes, permite buscar con una secuencia de una proteína empleando los métodos de HMM, que hoy por hoy son los más eficaces en el análisis de secuencias.
A los programas de BLAST, PSI-BLAST, PHI-BLAST.... se puede acceder desde el sitio del NCBI.
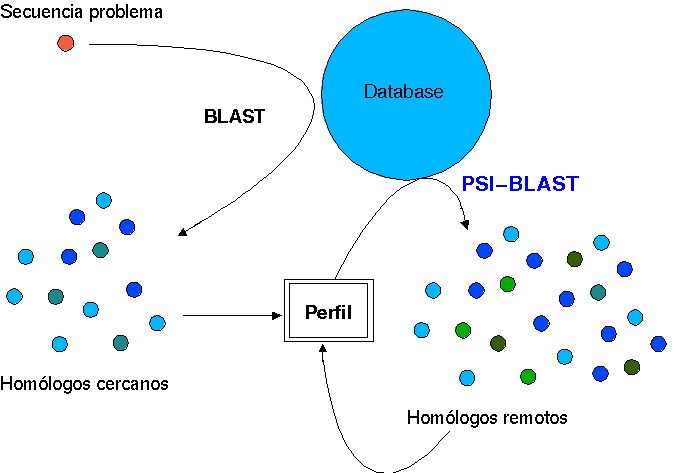
Además de este modo de funcionamiento básico a partir de una secuencia, PSI-BLAST también puede iniciarse con un alineamiento múltiple.
Ejecutarlo localmente, por línea de comandos, puede resultar un poco tedioso, y resulta más sencillo utilizar el servidor del NCBI, que además ofrece algunas ventajas: por ejemplo, permite seleccionar cuáles de los homólogos deberán ser utilizados para construir el perfil.
Ejemplo (el que se sigue en el tutorial
del NCBI)
>gi|2501594|sp|Q57997|Y577_METJA PROTEIN MJ0577
MSVMYKKILYPTDFSETAEIALKHVKAFKTLKAEEVILLHVIDEREIKKRDIFSLLLGVAGLNKSVEEFE
NELKNKLTEEAKNKMENIKKELEDVGFKVKDIIVVGIPHEEIVKIAEDEGVDIIIMGSHGKTNLKEILLG
SVTENVIKKSNKPVLVVKRKNS
PHI-BLAST (Pattern-Hit Initiated BLAST): permite realizar búsquedas que son mezcla de búsquedas de patrones y alineamiento de secuencias. Se inicia con una secuencia y un patrón (que presenta dicha secuencia y que es característico de dicha familia). Lo que hace es buscar todas aquellas secuencias que presenten ese patrón, pero para descartar aquellas ocurrencias del patrón que se producen al azar (especialmente con patrones cortos), además comprueba que las regiones vecinas al patrón sean parecidas entre las secuencias.
Instalación local
hmmbuild => hmmcalibrate => hmmsearch

Familias
de proteínas. Clasificación de proteínas.
Hasta ahora, en los apartados anteriores, en las que hemos explicado los conceptos de patrones y perfiles, hemos utilizado frecuentemente el concepto de familia de proteínas. De hecho hemos descrito dos bases de datos (Prosite y Pfam) que tienen como uno de sus principales objetivos clasificar las proteínas.
También hemos hablado de cómo evolucionan las proteínas y de que cuando dos proteínas tienen un origen evolutivo común decimos que son "homólogas". Hemos visto la utilidad de las herramientas del tipo HMMer o PSI-BLAST para identificar homologías remotas.
Sin embargo, el concepto
de homólogos se queda corto cuando queremos clasificar las proteínas.
A
lo largo de la evolución, a través de procesos de duplicación
génica y divergencia, y también mediante el barajado de dominios,
aparecen nuevas subfamilias de proteínas, con nuevas funciones.
Ejemplo hipotético de evolución de una proteína
Supongamos que en organismo ancestral se produce una duplicación de un hipotético gen que codifica para una proteína de secuencia:
A T F Y A G C D E L
A T F Y A G C D E LA T F Y A G C D E L
A T F Y A G C D E L (secuencia original)A L F Y A G C E E L (secuencia uno)A S Y Y A G C D E I (secuencia dos)
A T F Y A G C D E L (secuencia original)A L F Y A G C E E L (secuencia uno)A S Y Y A G G D E I (secuencia dos)
1 2 3 4 5 6 7 8 9 10A L F Y A G C E E L (secuencia original uno)A L F Y A G C E E LA I F R A G C E E TA I F R A G C E E LA V F Y A G C E E LA S Y Y A G G D E I (secuencia original dos)A S Y Y A G G D E IA T Y Y D G G D E IA T Y L A G G D E IA S R L A G G D E IA S Y Y A G G D E I
La situación actual. "Lo que vemos en las bases de datos".
La situación actual encaja bastante bien con ese modelo hipotético: observamos que en un grupo de proteínas homólogas en el que hay proteínas que tienen funciones distintas, las que tienen una misma función son más parecidas entre sí que con respecto a las otras.
Pero para describir las
relaciones entre las proteínas vemos que se nos queda corto el concepto
de homólogos, por lo que a veces hablamos en términos de
superfamilias,
familias y subfamilias y otras veces hablamos de ortólogos
y parálogos.
|
El interés de analizar la organización en familias de las proteínas
El objetivo más frecuente cuando estudiamos una proteína es llegar a conocer su función y averiguar cómo se las apaña para llevarla a cabo. Como hemos visto, conocer cuáles son sus homólogos nos puede ayudar, pero también es importante conocer cuáles pertenecen a su misma familia o subfamilia, de modo que, por ejemplo, podamos encontrar una correlación entre la conservación de determinados residuos en la subfamilia y características funcionales específicas de ésta. O por ejemplo, una correlación en la organización de dominios.
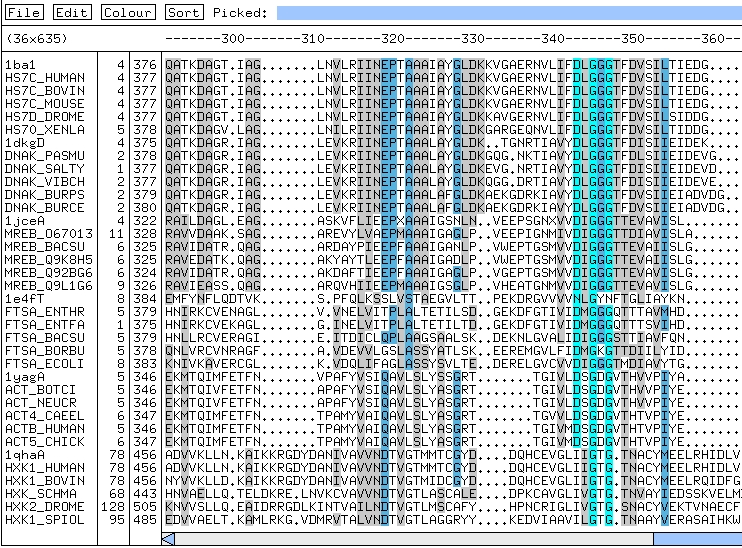
Alineamiento de proteínas de unión a ATP.
Algunos residuos están conservadas en todas las familias mientras
que otros varían mucho.
Y otros presentan un patrón de conservación
dependiente de cada familia.
En la superfamilia hay: chaperones (dnak), proteínas
implicadas en la formación del septo bacteriano (ftsA, mreB), hexokinasas
(hxk), actina (act)...
La forma más frecuente de hacer este tipo de análisis es construyendo un árbol filogenético (esto lo veréis otro día). El problema de los árboles es que uno tiene que buscar los homólogos, alinearlos, construir el árbol... y además de que esto puede llevar bastante tiempo, a veces los árboles resultantes no son buenos, especialmente si hay proteínas demasiado divergentes o si hay dominios no homólogos en las proteínas que intentamos alinear. Además, si queremos comparar dos genomas y ver qué funciones tiene uno y cuáles el otro (quizás intentándolo correlacionarlo con características fenotípicas de los organismos) debemos conocer las relaciones de ortología. Y esto no podemos hacerlo manualmente construyendo árboles para tantos genes. Por eso (entre otras razones) existen numerosas bases de datos y métodos para estudiar la organización de las familias de proteínas.
Las distintas bases de datos y los distintos métodos afrontan el problema de forma diferente, persiguiendo diversos objetivos. Unas aproximaciones tratan de encontrar grupos de ortólogos. Otras aproximaciones, grupos de homólogos. Etcétera.
En esta clase describiremos
algunas bases de datos de clasificación de proteínas y los
métodos que se emplean para construirlas.
Estas dos bases de datos ya las hemos explicado en la clase anterior. Se construyen a partir de perfiles-HMM una, y a partir de patrones y perfiles simples la otra.
(http://www.blocks.fhcrc.org/)
Podríamos dar toda una clase sobre las cosas que se pueden hacer
con BLOCKS, pero sólo daremos una visión general. Existe
un útil tutorial en BLOCKS.
Esta base de datos se construye a partir de familias descritas en InterPro
(ver más adelante) y Prints (ver más adelante). A partir
de los alineamientos de las proteínas de estas familias, se buscan
motivos
que estén conservados y que no presenten inserciones ni deleciones
(bloques; blocks). En un segundo paso, se determina cuál es el mejor
conjunto de motivos (o bloques) que definen a las proteínas de la
familia. Estos motivos se corresponden con sitios activos, sitios de unión
de substratos y cofactores y sitios con importantes implicaciones estructurales.
Esta clasificación tiene algunas ventajas porque permite que
fácilmente veamos cuáles son las pequeñas zonas conservadas
que son características de una familia. También es interesante
para determinar si una proteína de la familia carece de alguno de
los motivos.
Lo mejor de BLOCKS es el interfaz web que ofrece, permite realizar múltiples consultas, ver los perfiles de los motivos, construir árboles, buscar con los motivos en otras bases de datos...
Tour: buscaremos por "keyword", introduciendo "cytosine and methylase", gracias a lo cual obtendremos la entrada IPB001525 (se corresponde con la entrada IPR001525 de InterPro). Si hacemos click en ella veremos: una serie de motivos IPB001525A, IPB001525B, IPB001525C, IPB001525D, IPB001525E y IPB001525F. Podemos ver la longitud de cada uno y algunas otras características.
Por otra parte podemos ver la distribución de los motivos en todas las proteínas (pinchando en Block Map).
IPB001525: C-5 cytosine-specific DNA methylaseTambién podemos ver gráficamente los perfiles para cada motivo, pinchando en "Logos".
6 distinct blocks in 158 sequences
MTA1_ARTLU|P31974 ( 521) -A-----BB-CCC---DDD--------------------------EE-F-----
MTB6_BACSP|P43420 ( 315) A-----BB-CCC--DDD----------EE-F--
MTB1_BACBR|P34905 ( 374) -A------BB-CCC--DDD-----------EE-F----
MTD2_HERAU|P25265 ( 354) -A-----BB-CCC--DDD------------EE--F---
MTA1_RUEGE|P94147 ( 429) A-----BB-CCC--DDD---------------------EE-F--
(...)
Además, podemos utilizar una entrada de BLOCKS para buscar en bases de datos de secuencias, usando alguno de estos caminos:
Minimal length of reported alignments 4
Score cutoff is 5.6 Z score units (in the top 7.7e-05 percentile
of chance scores)
alignment Z-score expected number for
block 1 from:to
block 2 from:to length
searching 5000 blocks
IPB001525A 1 : 14 and IPB001525A
1 : 14 (14) score 100 (19.2 0.0e+00)
IPB001525A 2 : 14 and IPB001566A
3 : 15 (13) score 47 ( 6.2 1.3e-01)
IPB001525A 1 : 13 and IPB001614A
28 : 40 (13) score 45 ( 5.7 3.5e-01)
IPB001525B 1 : 16 and IPB001525B
1 : 16 (16) score 100 (21.1 0.0e+00)
IPB001525B 2 : 16 and IPB001532D
11 : 25 (15) score 41 ( 5.6 3.7e-01)
IPB001525C 9 : 27 and IPB000146D
3 : 21 (19) score 37 ( 5.7 3.5e-01)
IPB001525C 2 : 23 and IPB000352A
26 : 47 (22) score 36 ( 5.6 3.6e-01)
IPB001525C 1 : 28 and IPB001525C
1 : 28 (28) score 100 (31.8 0.0e+00)
IPB001525C 8 : 28 and IPB002857C
1 : 21 (21) score 43 ( 7.2 1.3e-02)
IPB001525D 1 : 27 and IPB001525D
1 : 27 (27) score 100 (31.2 0.0e+00)
IPB001525D 1 : 27 and IPB004000C
15 : 41 (27) score 29 ( 5.9 2.0e-01)
IPB001525E 1 : 16 and IPB001525E
1 : 16 (16) score 100 (21.1 0.0e+00)
IPB001525E 3 : 16 and IPB002584A
6 : 19 (14) score 46 ( 6.0 1.7e-01)
IPB001525E 1 : 16 and IPB002857F
16 : 31 (16) score 65 (12.1 0.0e+00)
IPB001525F 1 : 10 and IPB001525F
1 : 10 (10) score 100 (14.5 0.0e+00)
IPB001525F 1 : 10 and IPB002190D
20 : 29 (10) score 57 ( 6.5 5.5e-02)
En realidad lo que ocurre en este caso no es que haya dos familias relacionadas, sino que para definir a las metilasas éstas se ha utilizado una serie de motivos que abarcan varios dominios. La estructura de una de las metilasas es:
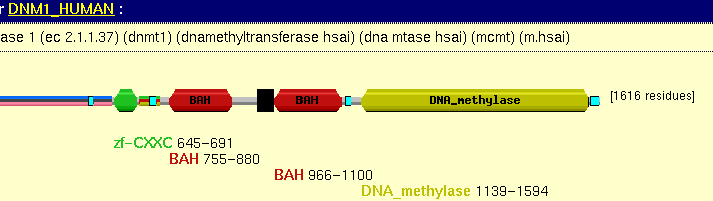
Sin embargo hay muchas otras proteínas que no son metilasas que presentan el dominio RING-Zn finger y para todas esas proteínas existe otra entrada en BLOCKs. Ambas entradas solapan.
(http://bioinf.man.ac.uk/dbbrowser/PRINTS/)
Aquí
tenéis un tutorial sobre PRINTS.
El enfoque de esta base de datos es similar al de BLOCKS. Agrupa las
proteínas en superfamilias, familias y subfamilias de acuerdo a
la presencia de conjuntos de pequeños motivos, a la "huella digital"
(fingerprint) que caracteriza a una familia de proteínas.
¿Cómo se construye la base de datos?: a partir de alineamientos
múltiples se derivan automáticamente perfiles para motivos
conservados. Con éstos se realizan búsquedas en las bases
de datos de secuencias. Con las nuevas proteínas se mejoran los
motivos, se construyen de nuevo perfiles para cada motivo y se realizan
nuevas búsquedas. Finalmente, se obtiene para la familia en cuestión,
una lista de motivos que caracterizan de forma óptima a la familia
y una lista de las proteínas que los presentan, indicando qué
proteínas presentan todos los motivos y cuáles sólo
algunos.
Un par de ejemplos de fingerprints: FASRECEPTOR
y IL1BCENZYME.
También existe un enlace para ver la estructura tridimensional
y dónde se localizan los motivos del 'fingerprint'.
Se pueden realizar búsquedas con FPScan
para determinar a qué fingerprints se parece una determinada secuencia.
Aquí
tenéis un ejemplo, el resultado de buscar con swiss:RASH_HUMAN.
(http://smart.embl-heidelberg.de/;
mirror en http://smart.ox.ac.uk/)
[ Smart Modular Architecture Research
Tool
]
La versión 3.5, de febrero de 2003, contiene 654 dominios.
Los dominios se construyen de forma parecida a Pfam: se elaboran HMMs semilla
para determinados dominios y con esos HMMs se busca en las bases de datos
para anotar las proteínas.
Para cada dominio se muestra información de en qué especies
está presente, con qué otros dominios aparece, la localización,
enlaces a la estructura tridimensional, etcétera. Muy parecido a
Pfam.
Quizás la característica más particular de SMART es que pone el énfasis en definir los dominios móviles de eucariotas, es decir, aquellos dominios más promiscuos como SH2, SH3, dominio plecstrina, etcétera los cuales aparecen en muy diversas combinaciones. Además ofrece un servicio de búsqueda de proteínas según la organización de dominios que tengan. Por ejemplo, podemos buscar todas aquellas proteínas que tengan los dominios CARD y CASc pero que no tengan el dominio BIR.
La definición de los dominios puede variar de una base de datos
a otra. Por ejemplo, según Pfam hay 137
proteínas que tienen el dominio caspasa, mientras que en SMART son
sólo 127.
[http://prodes.toulouse.inra.fr/prodom.html]
Hay un tutorial en http://prodes.toulouse.inra.fr/prodom/2002.1/documentation/help.php.
También es una base de datos de dominios. Su peculiaridad más sobresaliente es que la clasificación se hace automáticamente, usando el programa MKDOM2. La idea es que la secuencia más corta (siempre y cuando no sea un fragmento de una proteína) se corresponderá con un dominio. Con esa secuencia se inicia un PSI-BLAST iterativo y con los segmentos homólogos de otras proteínas ya tenemos definido un dominio. Estos segmentos son "eliminados" de la base de datos. Los segmentos de esas proteínas que no sean homólogos permanecen en la base de datos. Con la siguiente secuencia más corta se inicia otra vez el proceso. Y así sucesivamente hasta que no queden secuencias en la base de datos.
En las últimas versiones también se generan los dominios a partir de los que ya están descritos en Pfam-A. Y también algunos dominios son corregidos manualmente por expertos. A partir de estos dominios se realizan búsquedas PSI-BLAST del mismo modo que con los dominios descritos automáticamente.
El resultado son 305.465 dominios (de un total de una base de datos de unas 400.000 secuencias). Y existen 108.076 familias de proteínas con al menos dos dominios.
Esta base de datos puede resultar útil en aquellos casos en que no exista una definición de un dominio dado en otras bases de datos como Pfam, Prosite o Smart.
ProDom-CG (CG: complete genome) es un subconjunto de ProDom para los proteomas correspondientes a genomas ya secuenciados.
ProDom es la fuente de información que se utiliza para construir
el suplemento Pfam-B.
[http://www.ebi.ac.uk/interpro/]
Hay documentación en: http://www.ebi.ac.uk/interpro/user_manual.html?
Hemos visto que son muchas las bases de datos de dominios y motivos, que cada una describe distintos niveles de la organización de las proteínas (y muchas veces de una forma que no es consistente), que cada una usa distintos métodos... por eso se decidió crear InterPro, que es una base de datos que se construye a partir de Pfam, SMART, Prosite, Prints, ProDom y TIGRFams.
| Database | Version | Entries |
| SWISS-PROT | 41.0 | 122564 |
| PRINTS | 35.0 | 1750 |
| TrEMBL | 23.0 | 830525 |
| Pfam | 8.0 | 5193 |
| PROSITE patterns | 17.37 | 1605 |
| PROSITE preprofiles | N/A | 150 |
| ProDom | 2002.1 | 1021 |
| InterPro | 6.0 | 7751 |
| Smart | 3.4 | 654 |
| TIGRFAMs | 2.1 | 1614 |
La versión de InterPro 6.0 contiene 7751 entradas (1748 dominios,
5841 familias, 147 'repeats' y 15 sitios de modificación post-transduccional).
Hay 3.408.359 conexiones entre las 953.089 secuencias de Swiss-Prot
+ TrEMBL y las 7751 entradas de InterPro.
El 87% de las proteínas de Swiss-Prot (y el 75% de las de TrEMBL)
tienen al menos una conexión con InterPro.
|
Ejemplo: el dominio CARD.
En su entrada se indica que hay 117 proteínas que tienen este dominio.
También se indica que los dominios correscpondientes en Pfam, Prosite
y Smart (PF00619, PS50209 y SM00114) contienen 82, 115 y 72 proteínas,
respectivamente.
También se muestra una descripción de la función
de este dominio.
En el enlace "overview" (y en "...sorted by name" y en "detailed") se muestra gráficamente en qué regiones de cada una de las 117 proteínas aparece cada uno de los dominios Pfam, Prosite y Smart. También se muestra qué otros dominios presentan cada una de las 117 proteínas. Vemos que entre esas proteínas algunas presentan el dominio "proteína kinasa", otras el dominio "caspasa", etcétera. En "detailed" se muestra lo mismo pero más ampliamente. Por ejemplo, en la parte de "overview" vemos esto para la proteína RIK2_HUMAN:
![]()
Vemos que la proteína PIAP_PIG tiene varios dominios. Los dos primeros (los azules) son "BIR repeats" (si ponemos el ratón encima aparece una etiqueta que lo indica), luego está el dominio CARD y luego el dominio Zn-finger de tipo RING.
Y en "detailed" vemos esto:

Vemos que en el caso de la proteína PIAP_PIG el dominio CARD se encuentra con los tres marcadores: Pfam, Prosite y Smart (no ocurre lo mismo con todas las proteínas que según interPro tienen este dominio). Las repeticiones BIR se encuentran con 4 marcadores (PF0065, PS01282, PS50143 y SM00238). Los dominios Zn-finger se encuentran con tres marcadores. Al lado de cada marcador vemos con qué entrada de InterPro se corresponde.
En el enlace "table", vemos qué regiones de cada una de las 117 proteínas se corresponden con cada una de las "signatures".
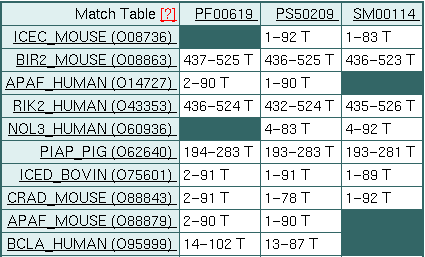
En la tabla además vemos cuáles de las "signatures" (marcadores) aparecen en cada proteína. En PIAP_PIG aparecen las tres, pero en NOL3_HUMAN, por ejemplo, no aparece el de Pfam. También se indica mediante las letras "T", "F" y "?", si la relación es correcta (T, true), incorrecta (F, false) o no se sabe (?).
El caso de PIAP_PIG: ¿qué función podría
tener?. Esta proteína está anotada en Swiss-Prot
como un posible inhibidor de la apoptosis. El dominio CARD es un dominio
de interacción entre proteínas. Normalmente interacciona
con otros dominios CARD, los cuales están presentes en caspasas,
kinasas, etc. (las caspasas son las principales ejecutoras del plan apoptótico
de la célula). Por otra parte, el dominio BIR (que está constituido
por varias repeticiones BIR) confiere resistencia a apoptosis. Además,
BIR normalmente aparece asociado al dominio Zn-RING finger, el cual está
implicado en interacciones proteína-proteína.
De forma rápida hemos podido hacernos una idea de la función
de esta proteína y de qué dominios tiene y qué rol
puede que desempeñen. Posiblemente, de confirmarse que esta proteína
es inhibidora de la apoptosis, lo haga interfiriendo en la maquinaria apoptótica,
quizás uniéndose a ella a través del dominio CARD,
y usando el Zn-RING finger para alguna otra interacción. Este tipo
de dedos de Zinc están asociados a muchos procesos celulares, como
por ejemplo a la ubiquitinación (etiquetado de proteínas
para su destrucción), ya que puede interaccionar con enzimas ubiquitinadoras,
pero no sabemos si éste es el caso.
Clasificación jerárquica en InterPro
Por ejemplo, para las proteínas kinasas existe esta jerarquía:

P.e. el IPR00719 (el nodo superior) está
definido por un dominio de ProDom (PD000001), también por uno de
Pfam (PF00069) y por otros dos: un patrón y un perfil de Prosite
(PS00107 y PS50011).
P.e. el nodo de las "Tyrosine protein kinases", el IPR001245,
está definido por una entrada de Prints (PR00109), otra de Prosite
(PS00109) y otra de SMART (SM00219).
etcétera.
[http://www.ncbi.nlm.nih.gov/COG/]
La documentación de COGs se encuentra en este
enlace.
COGs se refiere a "clusters of orthologous groups", es decir a grupos de genes ortólogos. Su objetivo es clasificar en tales grupos las proteínas de aquellos microorganismos de los que se conoce el genoma al completo. En la última versión había 43 genomas.
En este momento están diseñando una nueva versión de COGs que también será extensible a organismos pluricelulares como Arabidopsis thaliana, Caenorhabditis elegans, Drosophila melanogaster o Homo sapiens.
Clasificar las proteínas en grupos de ortólogos es muy útil para predecir la función de las mismas, ya que ésta se suele conservar en los ortólogos y entonces, conociendo la función de al menos una de las proteínas del grupo, podemos saber cuál es la función de las otras.
Además, para comparar genomas también es necesario
conocer las relaciones de ortología, ya sea para comparar el contenido
de los genomas o para comparar su organización.
|
Identifiación de ortólogos basada en best bidirectional hits (BBHs) La idea del método de 'best bidireccional hits (BBHs)' o mayores parecidos en las dos direcciones es que si una proteína de un genoma es la más parecida de otra en otro genoma, y viceversa (bidireccional), entonces es muy probable que ambas sean ortólogos. Por ejemplo, supongamos que tenemos dos genomas X e Y, y en cada uno de ellos hay dos proteínas homólogas A1 y A2 que proceden de una duplicación ancestral en un genoma 0 (A en la figura). Si ninguno de los dos genomas X e Y sufre una deleción, el método funcionará correctamente (caso B). Si se produce una deleción dependiendo de a qué genes afecte el método funcionará bien (C) o mal (D).
|
|
El sitio web de COGs ofrece muchas herramientas para consultar los resultados.
Se pueden buscar COGs por "palabras clave", por representación filogenética (p.e. obtener todos aquellos COGs en los que hay arqueas pero no bacterias), por clase funcional, por ruta metabólica, etcétera.
Una vez identificado un COG de interés (ejemplo) podemos ver qué función tiene o qué genomas están representados en él.
En COGs también existen herramientas para comparar genomas.
Por ejemplo viendo su organización estructural, como en el ejemplo
anterior. También hace un análisis
de componentes principales basado en la co-ocurrencia
de los genomas en los COGs.
Por otra parte, si queremos clasificar una secuencia podemos utilizar el programa COGNITOR (ayuda). Podemos buscar con swiss:DNAK_ECOLI.
>DNAK_ECOLI|P04475|Chaperone protein dnaK (Heat shock protein 70) (Heat shock 70 kDa protein) (HSP70).
GKIIGIDLGT TNSCVAIMDG TTPRVLENAE GDRTTPSIIA YTQDGETLVG QPAKRQAVTN
PQNTLFAIKR LIGRRFQDEE VQRDVSIMPF KIIAADNGDA WVEVKGQKMA PPQISAEVLK
KMKKTAEDYL GEPVTEAVIT VPAYFNDAQR QATKDAGRIA GLEVKRIINE PTAAALAYGL
DKGTGNRTIA VYDLGGGTFD ISIIEIDEVD GEKTFEVLAT NGDTHLGGED FDSRLINYLV
EEFKKDQGID LRNDPLAMQR LKEAAEKAKI ELSSAQQTDV NLPYITADAT GPKHMNIKVT
RAKLESLVED LVNRSIEPLK VALQDAGLSV SDIDDVILVG GQTRMPMVQK KVAEFFGKEP
RKDVNPDEAV AIGAAVQGGV LTGDVKDVLL LDVTPLSLGI ETMGGVMTTL IAKNTTIPTK
HSQVFSTAED NQSAVTIHVL QGERKRAADN KSLGQFNLDG INPAPRGMPQ IEVTFDIDAD
GILHVSAKDK NSGKEQKITI KASSGLNEDE IQKMVRDAEA NAEADRKFEE LVQTRNQGDH
LLHSTRKQVE EAGDKLPADD KTAIESALTA LETALKGEDK AAIEAKMQEL AQVSQKLMEI
AQQQHAQQQT AGADASANNA KDDDVVDAEF EEVKDKK
[http://protomap.cornell.edu/]
Una introducción
y un tour por
ProtoMap.
| ProtoMap es una base de datos que ofrece una clasificación
jerárquica del espacio de secuencias. El método es totalmente
automático.
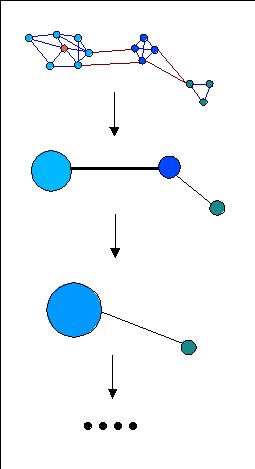
La clasificación se realiza en función de las distancias entre las secuencias, en función de cuánto se parecen. Se realiza una búsqueda mediante Smith & Waterman, BLAST y FASTA para cada una de las secuencias de Swiss-Prot+TrEMBL. De este modo se obtiene una medida de la distancia entre todas las proteínas. Estos resultados se representan mediante un grafo en el que los nodos son las proteínas y los nodos están unidos por arcos cuando alguno de los métodos ha encontrado un parecido. El peso de los arcos viene dado por el e-value asociado a tal parecido de secuencia. En este grafo las proteínas que se parezcan más estarán
más cerca entre sí (ejemplo). El
objetivo ahora es encontrar un algoritmo de clustering (agrupamiento)
capaz de identificar automáticamente los grupos de secuencias que
existen en dicho mapa.
|
 |
-la propia limitación de los métodos de comparación entre pares de secuencia, que tienen un menor poder de discriminación de homologías remotas. Sin embargo la comparación entre pares de secuencias tiene la ventaja de que ofrece una medida de la distancia evolutiva entre las secuencias (no así la comparación entre un perfil y una secuencia) lo cual permite aplicar el método de agrupamiento automático.
-la propiedad transitiva de la homología entre las proteínas
se aplica sin tener en cuenta la naturaleza multidominio de las mismas,
por lo que en un mismo cluster puede haber proteínas que no tengan
un mismo origen.
Interfaz web:
Se puede clasificar una nueva secuencia (realizando una búsqueda)
o bien se puede navegar por la jerarquía
de clusters, buscando por "keywords", "accession number", etc. Se puede
seleccionar también el nivel al que queremos acceder, desde 1e-0
hasta 1e-100, desde grupos más grandes a grupos más pequeños.
Ejemplo: swiss:RASH_HUMAN.Buscando con el identificador de swiss-prot averiguamos que esta proteína, en el nivel 1e-0, se encuentra en el cluster 18, que contiene 976 proteínas. También vemos con qué otros clusters tiene arcos el nodo de rash_human.
Si vamos al cluster 18 veremos qué proteínas hay en él, su función, su origen filogenético (virus, eucariotas, bacterias, etc). También vemos qué patrones y perfiles de Prosite están presentes en las proteínas del grupo.También podemos:
Cuestión práctica: ¿Hay alguna relación evolutiva entre la proteína ras/p21 de humanos y el factor de elongación de la traducción EF-Tu de E. coli?
- ver el alineamiento mútliple.
- ver los constituyentes de mayor orden ("higher order constituens"), donde podemos ver a partir de qué subgrupos se ha constituido este cluster (es decir, los grupos que había a umbrales más restrictivos).
- Y también podemos ver con qué otros clusters tienen relaciones (arcos, parecidos de secuencia) las proteínas de este grupo ("possibly related clusters"). Allí veremos los grupos vecinos ordenados de acuerdo a la "quality" que no es más que la media geométrica de los e-values, calculada como se ha mencionado antes. También vemos cuántos arcos hay entre los grupos y el tamaño de los grupos. Es muy útil para ver con qué otras familias está relacionada una familia determinada.
- En el servidor antiguo de ProtoMap sólo se aplicaba el método sobre Swiss-Prot por lo que se ha quedado un poco anticuado, pero tenía la ventaja de que permitía consultar el origen del grupo en forma de árbol, de forma que podíamos ver qué grupos se habían ido uniendo según se relajaba el umbral y qué proteínas había en cada uno, como en esta figura.
Estas son sus secuencias:
>RASH_HUMAN|P01112|Transforming protein P21/H-RAS-1 (C-H-RAS).
MTEYKLVVVG AGGVGKSALT IQLIQNHFVD EYDPTIEDSY RKQVVIDGET CLLDILDTAG
QEEYSAMRDQ YMRTGEGFLC VFAINNTKSF EDIHQYREQI KRVKDSDDVP MVLVGNKCDL
AARTVESRQA QDLARSYGIP YIETSAKTRQ GVEDAFYTLV REIRQHKLRK LNPPDESGPG
CMSCKCVLS
>EFTU_ECOLI|P02990|Elongation factor Tu (EF-Tu) (P-43).
SKEKFERTKP HVNVGTIGHV DHGKTTLTAA ITTVLAKTYG GAARAFDQID NAPEEKARGI
TINTSHVEYD TPTRHYAHVD CPGHADYVKN MITGAAQMDG AILVVAATDG PMPQTREHIL
LGRQVGVPYI IVFLNKCDMV DDEELLELVE MEVRELLSQY DFPGDDTPIV RGSALKALEG
DAEWEAKILE LAGFLDSYIP EPERAIDKPF LLPIEDVFSI SGRGTVVTGR VERGIIKVGE
EVEIVGIKET QKSTCTGVEM FRKLLDEGRA GENVGVLLRG IKREEIERGQ VLAKPGTIKP
HTKFESEVYI LSKDEGGRHT PFFKGYRPQF YFRTTDVTGT IELPEGVEMV MPGDNIKMVV
TLIHPIAMDD GLRFAIREGG RTVGAGVVAK VLSSi queréis podéis probar con BLAST para determinar esta cuestión. Pero os adelanto que el mejor alineamiento local que obtendréis será muy pequeño y poco significativo (=no distinguible de un parecido al azar entre el millón de proteínas conocidas):¿Podríamos haber detectado la relación evolutiva entre rash_human y EF-Tu de ecoli usando Pfam?
ras : ESRQAQDLARSYGIPYI
eftu: QTREHILLGRQVGVPYILos perfiles y los HMMs permiten determinar relaciones evolutivas distantes porque incorporan información precisa de la familia en cuestión, como por ejemplo que los residuos X e Y sean un Trp y una Lys, dando más importancia a la conservación de éstos que a la de otros.
1º. Id a Pfam y pinchad en el enlace de 'protein search'.
2º. Pegad la secuencia de rash_human y poned el 'E-value cutoff level' a 100. Obtendréis esto.
MTEYKLVVVG AGGVGKSALT IQLIQNHFVD EYDPTIEDSY RKQVVIDGET CLLDILDTAG3º. Haced lo mismo con eftu_ecoli en otra ventana. Obtendréis esto.
QEEYSAMRDQ YMRTGEGFLC VFAINNTKSF EDIHQYREQI KRVKDSDDVP MVLVGNKCDL
AARTVESRQA QDLARSYGIP YIETSAKTRQ GVEDAFYTLV REIRQHKLRK LNPPDESGPG
CMSCKCVLSSKEKFERTKP HVNVGTIGHV DHGKTTLTAA ITTVLAKTYG GAARAFDQID NAPEEKARGI¿Qué conclusión podéis sacar? ¿Es capaz de detectar la relación evolutiva? ¿Sale el dominio característico de cada una de estas proteínas en la lista de 'matches' del otro? ¿Con qué E-value? ¿Son iguales estos E-values? ¿Por qué?
TINTSHVEYD TPTRHYAHVD CPGHADYVKN MITGAAQMDG AILVVAATDG PMPQTREHIL
LGRQVGVPYI IVFLNKCDMV DDEELLELVE MEVRELLSQY DFPGDDTPIV RGSALKALEG
DAEWEAKILE LAGFLDSYIP EPERAIDKPF LLPIEDVFSI SGRGTVVTGR VERGIIKVGE
EVEIVGIKET QKSTCTGVEM FRKLLDEGRA GENVGVLLRG IKREEIERGQ VLAKPGTIKP
HTKFESEVYI LSKDEGGRHT PFFKGYRPQF YFRTTDVTGT IELPEGVEMV MPGDNIKMVV
TLIHPIAMDD GLRFAIREGG RTVGAGVVAK VLS
| volver al índice |