Análisis de secuencias:
motivos y perfiles
Parte práctica
Federico Abascal
En esta práctica veremos tres casos prácticos, cada uno
de los cuales intenta ilustrar alguno de los conceptos de "perfiles",
"familias" y "dominios".
Los casos que veremos serán los de las proteínas:
-
(Perfiles) swiss::YD33_MYCTU,
de Mycobacterium tuberculosis, que está anotada como: "Hypothetical
protein Rv1333".
-
(Familias) Proteína del gen
gcsf
de Bos taurus, anotada como: "Granulocyte colony-stimulating
factor precursor (G-CSF)".
-
(Dominios) swiss::ICE9_HUMAN,
de Homo sapiens; esta proteína es la
precursora de la caspasa-9.
1) YD33_MYCTU,
de
Mycobacterium
tuberculosis, que está anotada como: "Hypothetical protein
Rv1333".
En este ejercició intentaremos ilustrar que los métodos
basados en perfiles son capaces de detectar más homólogos
que los métodos simples como p.e. BLAST.
En este caso intentaremos proponer una posible función para esta
proteína que hemos obtenido de Swiss-Prot.
a) Primero obtendremos la secuencia de esta proteína a partir
de su identificador en swiss-prot (YD33_MYCTU):
alternativa 1: abre el SRS de http://srs.ebi.ac.uk/
, pincha en "start a temporary project", selecciona la base de datos Swiss-Prot
y busca por "YD33_MYCTU". Selecciona la vista "FastaSeqs" (está
debajo del botón view) y dale a "view".
alternativa 2 (más rápida): abre
la página de Swiss-Prot de http://us.expasy.org/sprot/
y busca "YD33_MYCTU" ("quick search"). Aparecerá la entrada Swiss-Prot
de esta proteína. Al final de la página hay un enlace que
dice "Q10644 in FASTA format". Pincha en ese enlace.
La secuencia:
>sw|Q10644|YD33_MYCTU Hypothetical protein Rv1333.
MNSITDVGGIRVGHYQRLDPDASLGAGWACGVTVVLPPPGTVGAVDCRGGAPGTRETDLL
DPANSVRFVDALLLAGGSAYGLAAADGVMRWLEEHRRGVAMDSGVVPIVPGAVIFDLPVG
GWNCRPTADFGYSACAAAGVDVAVGTVGVGVGARAGALKGGVGTASATLQSGVTVGVLAV
VNAAGNVVDPATGLPWMADLVGEFALRAPPAEQIAALAQLSSPLGAFNTPFNTTIGVIAC
DAALSPAACRRIAIAAHDGLARTIRPAHTPLDGDTVFALATGAVAVPPEAGVPAALSPET
QLVTAVGAAAADCLARAVLAGVLNAQPVAGIPTYRDMFPGAFGS
b) Ahora podemos hacer una búsqueda BLAST para ver si
se conoce la función de alguno de los homólogos.
Servidores de BLAST: EMBL,
NCBI,
EBI.
Pasos a seguir usando el servidor de BLAST del EMBL:
Ve a la página de BLAST del EMBL, que está en: http://dove.embl-heidelberg.de/Blast2/.
program=blastp (para comparar proteínas con proteínas)
database=nrdb95 (contiene todas las proteínas
conocidas pero ha eliminado aquellas que se parecen más de un 95%).
filter=SEG
descriptions=250
alignments=100
Pincha en "Submit Query".
Resultados: podéis encontrarlos aquí.
(y los resultados que
obtendríamos si no usásemos el filtrado de SEG)
Cuestiones:
¿Qué proteínas aparecen? ¿Nos aportan
alguna información sus anotaciones? ¿Qué significan
las "X" que véis en los alineamientos? ¿Merece la pena usar
el filtrado en este caso?
c) Ya que la mayoría de los homólogos no
tienen una función descrita, podemos intentar buscar homólogos
remotos utilizando búsquedas con perfiles.
Existen muchas formas posibles de hacer esto. Por ejemplo:
podríamos obtener las secuencias de los homólogos que han
aparecido usando BLAST y construir un alineamiento múltiple. Con
este alineamiento podríamos construir un perfil o un perfil de tipo
HMM y hacer nuevas búsquedas en las bases de datos de secuencias.
Lo más sencillo es utilizar PSI-BLAST ya que él mismo
se encarga de obtener las secuencias, alinearlas y construir un perfil
para nuevas búsquedas.
Pasos a seguir usando el servidor de PSI-BLAST del NCBI:
Ve a la página inicial de BLAST del NCBI, que está en:
http://www.ncbi.nlm.nih.gov/BLAST/.
Allí ve a "Protein BLAST / PSI- and PHI-BLAST".
Pega la secuencia en el campo "Search".
Descriptions=500
Alignments=100
Format for PSI-BLAST -> with inclussion threshold = 1e-05 (=0.00001)
Dale a "BLAST!"
Al cabo de un rato pincha en "format" y en otra ventana aparecerán
los resultados de la primera ronda. Échales un vistazo y cuando
termines: selecciona las proteínas que quieres que se utilicen para
construir el perfil y pincha en "Run PSI-BLAST iteration 2".
Otra vez, al cabo de un rato, pincha en "format" y en otra
ventana aparecerán los resultados de la segunda ronda.
Y así sucesivamente.
Resultados de la primera
ronda, de la segunda y de la tercera
ronda.
Cuestiones:
¿Conseguimos encontrar más homólogos con
PSI-BLAST? ¿Son más claras sus anotaciones? ¿Qué
hipótesis acerca de la función de la proteína podríamos
proponer?
Por otra parte, en la tercera ronda, las proteínas que aparecen
al principio de la lista (que está ordenada según la puntuación)
tienen porcentajes de identidad de secuencia muy bajos con respecto a YD33_MYCTU,
sin embargo los e-values son muy buenos, y en cambio, las proteínas
que son más parecidas a YD33_MYCTU (mayor % de identidad) tienen
e-values peores. ¿Por qué?
d) Búsqueda en Pfam.
Ahora veremos qué obtendríamos si comparásemos
la secuencia de YD33_MYCTU con los perfiles-HMM de Pfam.
Pasos a seguir:
Ve a la página de Pfam, que está en: http://www.sanger.ac.uk/Pfam/.
Ve "Protein Search". Una vez allí:
pega la secuencia.
E-value=10
Dale a "Search Pfam".
Resultados de la búsqueda en Pfam.
Cuestiones:
¿Cuántos dominios tiene esta proteína?
¿En qué zona de la proteína se localizan? ¿Qué
función tienen? ¿Qué podemos decir de la función
de la proteína?
Cuestiones respecto al dominio "peptidasa_T4": ¿cuántas
proteínas presentan este dominio? ¿con qué otros dominios
puede aparecer asociado?
2) Proteína del gen gcsf
de Bos taurus (Granulocyte colony-stimulating factor precursor)
En esta práctica intentaremos ilustrar el papel de las familias
de proteínas en el análisis de secuencias.
a) Primero: obtención de la secuecia aminoacídica correspondiente
a este gen:
Pasos a seguir:
Ve al servidor de SRS del EBI: http://srs.ebi.ac.uk/.
Dale a "Start a Temporary Project".
Selecciona algunas bases de datos de proteínas (aún no
sabemos en qué base de datos está). P.e. selecciona: Swiss-prot,
SpTrEMBL y TrEMBL (updates).
Pincha en "Query forms => Standard".
Busca por: "GeneName=gcsf" y "Organism name=Bos taurus".
Pincha en "submit query".
Y ahora lo mismo que antes: selecciona la "View" de tipo "FastaSeqs"
y pincha en "view".
La secuencia:
>sw|P35833|CSF3_BOVIN Granulocyte colony-stimulating factor precursor
(G-CSF).
MKLMVLQLLLWHSALWTVHEATPLGPARSLPQSFLLKCLEQVRKIQADGAELQERLCAAH
KLCHPEELMLLRHSLGIPQAPLSSCSSQSLQLTSCLNQLHGGLFLYQGLLQALAGISPEL
APTLDTLQLDVTDFATNIWLQMEDLGAAPAVQPTQGAMPTFTSAFQRRAGGVLVASQLHR
FLELAYRGLRYLAEP
b) Búsqueda PSI-BLAST con la secuencia de swiss::CSF3_BOVIN.
Podéis hacer la búsqueda PSI-BLAST vosotros mismos
(siguiendo los pasos del ejemplo anterior) o, para ahorrar tiempo, consultar
los resultados en este fichero.
Intenta comprender qué ha sucedido a lo largo de las distintas
rondas.
Cuestiones:
-¿Qué nos ha permitido averiguar PSI-BLAST?
-En la segunda ronda aparece una proteína con una anotación
diferente: "Q90YI0 (Q90YI0) Interleukin-6 precursor". Ésta tiene
una identidad del 20% con respecto a la "query", sin embargo tiene un e-value
bastante significativo: ¿por qué?
-¿Qué consecuencias tiene la inclusión de esta
nueva secuencia en el perfil?
-¿Por qué en la última ronda las proteínas
"interleukin..." tienen mejores e-values que las proteínas "granulocyte..."?
-Todas las proteínas que aparecen tienen un origen evolutivo
común, pero ¿realizan la misma función? ¿qué
subfamilias podéis identificar?
¿Cómo creéis que la existencia de subfamilias
con funciones distintas puede afectar a la predicción de función
a partir de homólogos?
c) Búsqueda en bases de datos de familias: Pfam e InterPro.
Búsqueda en Pfam.
Pasos a seguir:
repite los pasos del ejemplo anterior.
El resultado de la búsqueda es éste.
Cuestiones:
-Lee la documentación de Pfam correspondiente al dominio
de esta proteína. ¿Qué subfamilias se agrupan en esta
entrada?
-Por otra parte, en que el resultado de la búsqueda
vemos que tiene un parecido remoto (e-value de 4.3) con la familia IL-12.
¿Crees que este parecido refleja una origen común de ambos
grupos de proteínas (las que están en el grupo IL-6+gcsf+etcétera
y las del grupo IL10)? Formas de determinarlo: viendo el alineamiento múltiple,
viendo si las estructuras de las proteínas de ambos grupos se parecen,
etcétera.
Búsqueda en InterPro.
Breve introducción acerca de InterPro: hemos
visto que en Pfam se describe un grupo que contiene varias subfamilias.
En otras bases de datos de estas características (Prosite, SMART,
PRINTS, ProDom, TIGRFAMs...) los grupos pueden aparecer definidos a otros
niveles. Por ejemplo, puede ocurrir que un grupo de Pfam se corresponda
con varios grupos de PRINTS. Y puede ocurrir que un grupo de Prosite se
corresponda con varios de Pfam. Cada base de datos refleja la jerarquía
de relaciones en superfamilias-familias-subfamilias a un nivel distinto.
En InterPro se ponen en conjunto los datos de las distintas bases de datos.
Por ejemplo, para las proteínas kinasas existe esta jerarquía:

P.e. el IPR00719 (el nodo superior) está
definido por un dominio de ProDom (PD000001), también por uno de
Pfam (PF00069) y por otros dos: un patrón y un perfil de Prosite
(PS00107 y PS50011).
P.e. el nodo de las "Tyrosine protein kinases", el IPR001245,
está definido por una entrada de Prints (PR00109), otra de Prosite
(PS00109) y otra de SMART (SM00219).
etcétera.
Búsqueda con la secuencia de gscf
en InterPro
Pasos a seguir:
Ve a InterProScan, que está en: http://www.ebi.ac.uk/interpro/scan.html.
Pega la secuencia de la proteína gscf.
Escribe una dirección de e-mail cualquiera (da igual)
Y dale a "Submit job".
Cuestiones:
-¿Con qué entradas de InterPro tiene parecidos?
-De los dos parecidos que aparecen ¿qué jerarquía
hay entre ellos? P.e. ¿uno de los dos engloba al otro?
-En este caso: ¿Nos sirve la información de InterPro
para determinar a qué subfamilia pertenece la proteína?
3) ICE9_HUMAN,
de Homo Sapiens; esta proteína es la precursora de la caspasa-9.
a) obtenemos su secuencia:
esto lo podemos hacer como en el caso del primer ejemplo, a
partir de su identificador de Swiss-Prot: ICE9_HUMAN.
La secuencia es:
>ICE9_HUMAN
MDEADRRLLR RCRLRLVEEL QVDQLWDALL SRELFRPHMI EDIQRAGSGS RRDQARQLII
DLETRGSQAL PLFISCLEDT GQDMLASFLR TNRQAAKLSK PTLENLTPVV LRPEIRKPEV
LRPETPRPVD IGSGGFGDVG ALESLRGNAD LAYILSMEPC GHCLIINNVN FCRESGLRTR
TGSNIDCEKL RRRFSSLHFM VEVKGDLTAK KMVLALLELA QQDHGALDCC VVVILSHGCQ
ASHLQFPGAV YGTDGCPVSV EKIVNIFNGT SCPSLGGKPK LFFIQACGGE QKDHGFEVAS
TSPEDESPGS NPEPDATPFQ EGLRTFDQLD AISSLPTPSD IFVSYSTFPG FVSWRDPKSG
SWYVETLDDI FEQWAHSEDL QSLLLRVANA VSVKGIYKQM PGCFNFLRKK LFFKTS
b) hacemos BLAST:
El resultado sería éste.
Echa un vistazo a qué proteínas aparecen y al gráfico
en el que se muestran qué zonas son las que han alineado.
Cuestiones:
-¿Te dice algo el gráfico que indica qué
zonas han alineado? ¿Podrías relacionar lo que ves con la
presencia de distintos dominios en la proteína?
c) Búsqueda en Pfam:
El resultado sería éste.
Cuestiones:
-¿Cuántos dominios presenta esta proteína?
-¿Qué función tiene cada uno de estos dominios?
¿Qué sentido puede tener que estos dominios aparezcan juntos?
-Para cada uno de los dominios, mira la organización de dominios
de las proteínas, para ver con qué otros dominios pueden
asociarse. Pasos a seguir: Ve a "domain organisation", marca "full"
y luego dale a "Graphic".
*Cuestión: ¿con qué otros dominios se asocian?.
*Cuestión: sabemos que esta proteína interacciona
con apaf-1 (se dice en la entrada de Swiss-Prot).
Buscando con SRS en Swiss-Prot por
"description=apaf-1" + "organism name=Homo sapiens",
identificamos la entrada correspondiente a apaf-1, que es la de APAF_HUMAN.
¿cómo crees que se produce la interacción entre ambas
proteínas (caspasa-9 y apaf-1)?
*Cuestión: en la lista de parecidos también aparece
el dominio death,
aunque con un e-value malo, de 5.5. Intenta averiguar si ese parecido es
casual o refleja una relación de homología (opciones: consulta
las anotaciones de los dominios, a ver si se dice algo; o mira qué
tal es el alineamiento de la caspasa-9 con la semilla del dominio
death (con JalView se ve bastante bien), etc.
[CARD,
dominios;
caspase,
dominios]
Cuando hayas terminado trata de comprender...:
-...qué implicación tiene en el análisis de secuencias
que a lo largo de la evolución se haya producido tal barajado de
dominos.
-...qué consecuencias tiene a la hora de predecir la función
de las proteínas.
-...cómo puede afectarnos ese barajado de dominios a la hora
de construir alineamientos múltiples de proteínas.
d) Construcción de un perfil a partir de un alineamiento
múltiple y búsqueda con ese perfil:
Una vez que hemos comprendido que cuando queremos analizar
una proteína multidominio es mejor identificar sus dominios, generaremos
un perfil para un alineamiento múltiple de dominios CARD y haremos
una búsqueda con él. Usaremos el servidor Bioccelerator.
(Esto es equivalente a lo que hacemos con PSI-BLAST, pero
es un método un poco 'más mejor' y además vamos a
decirle nosotros a partir de qué alineamiento tiene que construir
el perfil)
Pasos a seguir:
1.- A partir de un alineamiento múltiple, por
ejemplo el del dominio CARD que podemos obtener en pfam (lo salvamos como
fasta),
tenemos que construir un perfil o PSSM (position specific scoring matrix).
Usamos el programa "ProfileWeight".
Una vez generada la matriz PSSM (el perfil) la guardamos en un fichero.
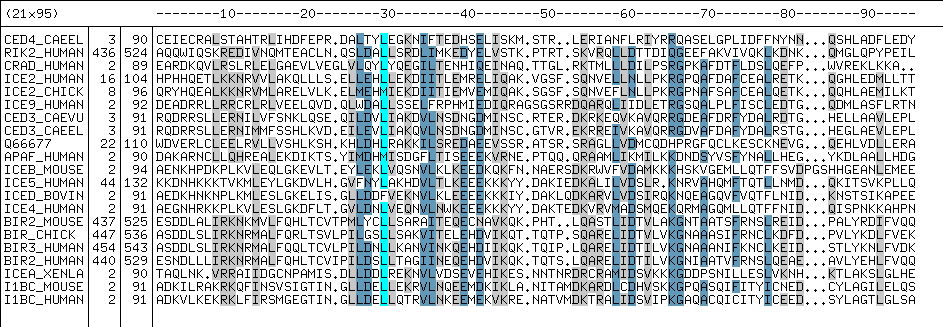
En la matriz podemos observar cómo aquellas secuencias más
divergentes tienen pesos mayores.
Sequence Weights:
1 CED4_CAEEL/3-90 100
2 RIK2_HUMAN/436-524 94
3 CRAD_HUMAN/2-89 92
4 ICE2_HUMAN/16-104 54
5 ICE2_CHICK/8-96 62
6 ICE9_HUMAN/2-92 83
7 CED3_CAEVU/3-91 56
8 CED3_CAEEL/3-91 58
9 Q66677/22-110 89
10 APAF_HUMAN/2-90 96
11 ICEB_MOUSE/2-94 79
12 ICE5_HUMAN/44-132 69
13 ICED_BOVIN/2-91 62
14 ICE4_HUMAN/2-91 65
15 BIR2_MOUSE/437-525 58
...etc.
2.- Seguidamente podemos buscar con ese perfil en una base
de datos de secuencias. Para ello pinchamos en el enlace "profilesearch".
Una vez allí, debemos cargar la matriz (en "upload file"). Luego
establecemos las penalizaciones por abrir y extender un gap a 11 y 1, respectivamente
(por poner un ejemplo).
3.- Finalmente, buscamos y obtenemos esto.
Cuestiones:
-Compara el resultado de la búsqueda
con el perfil al resultado de buscar con
BLAST con una de las secuencias del alineamiento, con ICE2_HUMAN:
>ICE2_HUMAN/16-104
HPHHQETLKKNRVVLAKQLLLSELLEHLLEKDIITLEMRELIQAKVGSFSQNVELLN
LLPKRGPQAFDAFCEALRETKQGHLEDMLLTT
-¿Qué método es capaz de detectar mayor cantidad de
homólogos?

