Especialmente en el caso de genomas
eucariotas, la primera fase del proceso de predicción de genes es
localizar zonas de secuencias repetidas, en las que es poco probable encontrar
elementos reguladores y secuencias codificantes. Las zonas de secuencias
repetidas pueden ser entonces descartadas a la hora de ejecutar otros tipos
de análisis, o enmascaradas, mediante la sustitución de los
caracteres que representan los cuatro nucleótidos por otro símbolo
arbitrario, por ejemplo "N" o "X".
Además de servir para descartar regiones
de menor interés desde el punto de vista funcional, el enmascaramiento
de secuencias repetidas facilita la búsqueda de secuencias similares
en bases de datos de secuencias con, por ejemplo, programas de la familia
de aplicaciones BLAST.
Normalmente, el enmascaramiento es realizado
de forma automática por muchos programas. En BLASTN,
por
ejemplo, el usuario puede seleccionar distintos tipos de filtro,
aunque la opción por defecto enmascara las denominadas regiones
de baja complejidad mediante un programa llamado DUST. Otras opciones en
BLASTN
permiten enmascarar secuencias repetidas humanas (LINEs y SINEs) o secuencias
especificadas por el usuario, mediante el uso de minúsculas, en
la secuencia usada como "query".
La búsqueda de genes o de productos génicos
homólogos es uno de los métodos más antiguos y más
usados para identificar secuencias codificantes y determinar la estructura
de genes. Este tipo de métodos se califican como extrínsecos
porque se basan en usar información procedente del estudio de otros
genomas. Son, por tanto, menos eficientes en Eucariotas que en Procariotas
dada la menor abundancia de información de especies evolutivamente
cercanas.
La familia de aplicaciones BLAST
incluye programas que permiten hacer diferentes tipos de búsquedas.
-
BLASTN permitiria identificar genes parecidos en bases de datos de secuencias
de DNA, usando la secuencia genomica como "query".
-
BLASTX traduce la secuencia "query" en las seis fases de lectura posibles
y hace busquedas en bases de datos de proteinas; esto permitiria identificar
productos genicos parecidos a los codificados por la secuencia genomica,
y serviria para identificar regiones codificantes (no intrones).
-
TBLASTX traduce la secuencia "query" en las seis fases abiertas de lectura
y hace busquedas en bases de datos de secuencias de DNA tambien traducidas
en la seis fases de lectura posibles; esto serviria para identificar secuencias
que codifican para productos parecidos a los que codifica la secuencia
"query".
Si se conoce, o se puede predecir, la secuencia del producto genico,
o parte de ella, otros programas permiten usar la secuencia de aminoacidos
como "query":
-
BLASTP busca proteinas parecidas en bases de datos de proteinas.
-
TBLASTN hace busquedas en bases de datos de secuencias de DNA traducidas
en la seis fases de lectura posible.
El programa Procrustes
está diseñado para intentar deducir la estructura de un gen,
en cuanto a intrones y exones, basándose en la secuencia de aminoácidos
de una serie de proteínas homólogas a las que supone está
codificada por el fragmento de DNA que se está analizando. La secuencia
de dichas proteínas homólogas tiene que ser facilitada por
el usuario del programa, que podría haberlas identificado mediante
búsquedas con BLASTX, por ejemplo. Si la secuencia aminoacídica
facilitada es la que codifica el fragmento de DNA, Procrustes es capaz
de reconstruir la estructura del gen con un 99% de exactitud.
La figura de abajo es un ejemplo de resultado obtenido con Procrustes,
en el que proteinas homólogas de varios organismos han sido alineadas
con una secuencia genómica de DNA, para predecir la organización
de intrones y exones.

Al igual que el análisis estadístico de la composición
del DNA, el análisis de señales o motivos también
se considera un método de tipo intrínseco. Estos métodos
se basan en la identificación de motivos de secuencia característicos
de los elementos que forman parte de los genes, tales como promotores,
codones de inicio y terminación, sitios de procesamiento del RNA
(en Eucariotas), terminadores de transcripción (en Procariotas),
etc.
El grado de conservación de cualquiera de esos motivos varía
considerablemente. Aquellos relativamente conservados pueden ser identificados
mediante búsquedas con secuencias consenso, que representan
la secuencia del motivo para una cierta mayoría de ejemplos. Fuzznuc
es un ejemplo de programa para buscar ocurrencias de una secuencia consenso
en otra secuencia. El programa permite especificar el número de
fallos (mismatches) que serían aceptables, así como
utilizar secuencias consenso ambiguas (por ejemplo, [ACG] significa A,
C o G).
Las secuencias consenso (incluso las ambiguas) tienen el problema
de que no contienen información de la frecuencia con que cada nucleótido
ocurre en cada posición. Dicha información puede ser expresada,
sin embargo, en forma de perfiles (profiles o position
weight matrices, PWMs), que son tablas en las que se recoje
la frecuencia con que cada nucleótido aparece en cada posición
a lo largo de la secuencia.
Una aproximación parecida es la de los algoritmos que usan
modelos
ocultos de Markov (Hidden Markov Models, HMM).
Muchos de los programas desarrollados recientemente para detectar los límites
entre exones e intrones (ver sección
3) usan esta estrategia.
Estos métodos hacen uso de información proviniente
de técnicas experimentales diseñadas para identificar productos
génicos de forma masiva. Ejemplos representativos de esta estrategia
son el uso de bases de datos de ESTs, de cDNAs completos y de secuencias
N-terminales de proteínas, que son utilizados para confirmar la
existencia de genes y para deducir la estructura genica.
Los ESTs (Expressed Sequence Tags) son secuencias cortas de DNA (de
200 a 500 pb), generadas mediante la secuenciación de los extremos
5' y 3' de clones de cDNA seleccionados al azar. Los clones de cDNA se
obtienen, a su vez, por transcripción reversa de RNA mensajero,
usando un oligo(dT) como primer, normalmente, lo que asegura que al menos
la secuencia del extremo 3' del RNA está incluida en el cDNA. Aunque
los cDNA así obtenidos suelen corresponder con fragmentos de RNA
mensajeros, la secuenciación de los extremos de los clones permite
la identificación, de una manera rápida, del repertorio de
genes expresados en una cierta condición, por un cierto tipo de
células.
Dado que las genotecas de cDNA se obtienen a partir de RNA mensajero
que ha sido ya procesado (maduro), los ESTs consisten en secuencias
exónicas. Dependiendo del grado de degradación del RNA original,
de la eficacia de la Transcriptasa Reversa y de la organización
y la longitud de cada gen, las secuencias de los ESTs pueden corresponder
a otros exones además de al terminal.
Por tanto, la identificación de ESTs que corresponden
con fragmentos de una secuencia de DNA genómico, primero, sirve
para detectar la presencia de un gen; y segundo, puede servir para deducir
la organización de sus exones e intrones.
La principal base de datos pública sobre ESTs es dbEST,
que es mantenida por el NCBI y que es, de hecho, una de las divisiones
de la base de datos GenBank.
La base de datos incluye un inventario
que se atualiza periódicamente y en el que se desglosa la composición
de las entradas por organismos. El 31 de Mayo
de 2002 había unas más de 11.800.000 entradas, de las
cuales 4,458,865 correspondían a ESTs humanos y 2,590,400 a ESTs
de ratón. La base de datos aumenta muy rápidamente. Las entradas
de la base de datos pueden ser recuperadas mediante búsquedas con
palabras clave, desde la página principal de dbEST.
A la hora de desarrollar el modelo de un gen, una secuencia genómica
se puede usar como "query" en búsquedas con BLASTN
restringidas a la base de datos de ESTs. El EBI mantiene una página
con enlaces a sitios web relacionados con ESTs (EST
Links).
Hay que tener en cuenta que el uso de los ESTs para detectar o modelar
genes tiene las siguientes limitaciones:
-
Como ya se ha mencionado, suelen representar sólo el extremo
3' del gen.
-
Son secuencias de regular calidad, que pueden tener errores (hasta el
3%, frente al 0.01% de secuencias genómicas humanas; Collins et
al 1998).
-
Pueden corresponder a secuencias intrónicas si la librería
de cDNA estaba contaminada con DNA genómico (Wolfsberg and Landsman,
1977)
-
Esta última posibilidad no es distinguible de los casos de splicing
alternativo, que se presume ocurre en un 35% de los genes humanos (Mironov
et al 1999).
cDNAs completos: dbEST almacena no sólo secuencias de ESTs, sino
secuencias de cDNA completos, cuyas secuencias pueden corresponder a la
práctica totalidad de los ARNm maduros que sirvieron de molde. Las
secuencias de cDNA completos pueden provenir de grupos experimentales que
han caracterizado genes individualmente. Sin embargo, existen también
una serie de proyectos a nivel mundial cuyos esfuerzos están orientados
a obtener sistemáticamente la secuencia completa de todas las especies
de RNA producidas por un organismo dado, mediante la secuenciación
completa de genotecas de cDNA. La catalogación de todos los RNAs
producidos por un organismo (o un tejido, o un tipo celular), junto con
los datos de expresión obtenidos en experimentos de gran escala
con DNA arrays, contribuirán a definir los TRANSCRIPTOMAS de cada
organismo.
A continuación se presenta una lista con enlaces a las
páginas web de algunos proyectos de secuenciación de cDNAs.
Por supuesto, la disponibilidad de secuencias completas de cDNA
simplifica enormemente el problema de mapear sobre el genoma la posición
de los intrones y exones de un gen. El alineamiento de un cDNA con su correspondiente
secuencia genómica sirve para identificar, de forma casi automática,
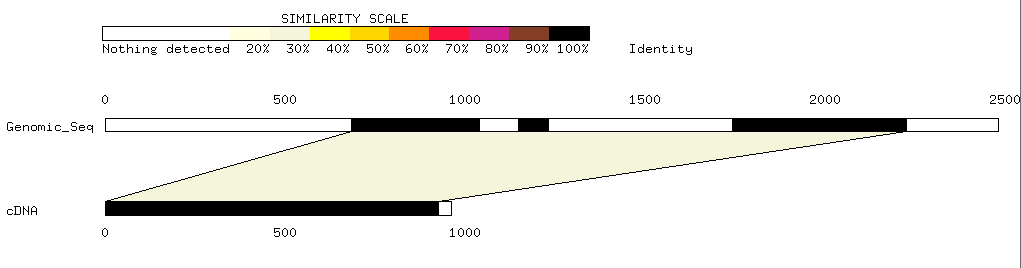
la localización de los exones y los intrones. Como ejemplo, la figura
de abajo muestra un esquema del alineamiento de un cDNA (GenBank Acc AL541035)
con su correspondiente secuencia genómica, realizado con el programa
SIM4
y visualizado con
LalnView.
El gen tiene tres exones, cuyas secuencias son mas de un 90% similares
a las correspondientes secuencias genómicas, y dos intrones. La
cola poli A del extremo 3' del cDNA, obviamente, no tiene correspondencia
en la secuencia genómica.
Por último, igual que la identificación de ESTs o cDNAs
que corresponden a una secuencia genómica es indicativo de la existencia
de un gen funcional, la identificación de proteinas cuya expresión
ha sido verificada experimentalmente y cuya secuencia está codificada
por una secuencia genómica, también demuestra la presencia
de un gen que se expresa.
En este sentido, las iniciativas para caracterizar el repertorio
de proteinas expresadas por un organismo, o PROTEOMA, ayudarán a
identificar nuevos genes o a confirmar su predicción. Si la secuencia
del extremo N terminal ha sido obtenida experimentalmente, dicha información
permite deducir el codon de iniciación de la fase abierta de lectura.
Como ejemplo, la identificacion de genes por búsqueda en un
base de datos de secuencias N-terminales de proteinas fue utilizada para
anotar el genoma de Escherichia coli [Science 277:1453 (1999)].
Una lista de proteinas de Escherichia coli cuyo extremo amino ha
sido confirmado experimentalmente, por secuenciación del extremo
amino terminal, puede encontrarse en EcoGene.
La lista incluye 850 proteínas de las cuales se puede obtener bibliografía
e información adicional, mediante enlaces a otras bases de datos.


